Voy a continuación a traduciros un interesante paper con fecha de junio de este año 2020. Los comentarios [entre corchetes] son anotaciones mías.
==================================================================================
La inmunidad protectora del coronavirus es de corta duración.
Autores
Arthur WD Edridge 1 , Joanna Kaczorowska 1 , Alexis CR Hoste 2 , Margreet Bakker 1 , Michelle Klein 1 ,Maarten F. Jebbink 1 , Amy Matser 3 , Cormac M. Kinsella 1 , Paloma Rueda 2 , Maria Prins 3,4 , Patricia Sastre 2 ,Martin Deijs 1 , Lia van der Hoek *
Afiliaciones
1. Laboratorio de Virología Experimental, Departamento de Microbiología Médica y Prevención de Infecciones,Instituto de Infección e Inmunidad de Amsterdam, Amsterdam UMC, Universidad de Amsterdam, Meibergdreef 15,1105 AZ Amsterdam, Países Bajos.
2. INGENASA, Inmunología y Genética Aplicada SA, Av. de la Institución Libre de Enseñanza, 39, 28037Madrid, España
3. Departamento de Enfermedades Infecciosas, Servicio de Salud Pública de Amsterdam, Nieuwe Achtergracht 100, 1018.WT Amsterdam Países Bajos.4. Amsterdam UMC, Universidad de Amsterdam, Departamento de Enfermedades Infecciosas, Infección y AmsterdamImmunity Institute, Amsterdam UMC, Universidad de Amsterdam, Meibergdreef 15, 1105 AZ Amsterdam,Los países bajos.
ABSTRACT
En la pandemia actual de COVID-19, una pregunta clave sin resolver es la duración de la inmunidad adquirida en individuos recuperados. La reciente aparición de SARS-CoV-2 impide un estudio directo sobre este virus, pero cuatro coronavirus humanos estacionales pueden revelar características comunes aplicables a todos los coronavirus humanos.
Monitoreamos sujetos sanos durante un período de 35 años (1985-2020), proporcionando un seguimiento total de 2473 personas/meses. Se determinó: a) el tiempo de reinfección por cada cepa de coronavirus, y b) la evolución en la disminución de anticuerpos contra estos coronavirus tras la infección y posterior recuperación. Como resultado se encontró una alarmante corta duración en la inmunidad protectora frente a dichos coronavirus. Ocurrieron reinfecciones frecuentemente transcurridos 12 meses tras la enfermedad, y hubo, para cada virus, una reducción sustancial en los niveles de anticuerpos en un tiempo tan temprano como 6 meses.
TEXTO PRINCIPAL
El SARS-CoV-2 es un nuevo coronavirus responsable de la pandemia en curso. Su rápida transmisión es probablemente causada por el hecho de que el virus entró en un ser humano muy desprotegido y por lo tanto altamente susceptible de ser infectado, combinado con la capacidad del propio virus para transmitirse durante la fase asintomática de la infección.
Como no hay intervenciones farmacéuticas disponibles actualmente, las políticas actuales para limitar la propagación del SARS-CoV-2 gira en torno a la contención, el distanciamiento social y la suposición de que los pacientes recuperados desarrollan inmunidad protectora. La duración de la protección afectará no solo el curso general de la pandemia actual, sino también el período posterior a la pandemia misma. Hasta la fecha no hay evidencia concreta de reinfección por SARS-CoV-2, ni tampoco ningún ejemplo de reinfección por SARS-CoV-1 o MERS-CoV; sin embargo, esto es probablemente debido a la reciente aparición de SARS-CoV-2 y a la poca escalada infectiva que tuvo el SARS-CoV-1 y MERS-CoV [que no llegaron a convertirse en pandemia]. En general todos suponen que sí puede ocurrir una reinfección por cualquier tipo de coronavirus, sin embargo, esta tesis se funda de momento en un único estudio experimental sobre individuos voluntarios en donde se usó un coronavirus cultivado (HCoV-229E) inoculado en intervalos de 12 meses. Por lo tanto, debemos señalar que la susceptibilidad a la reinfección natural por coronavirus no ha sido investigada hasta ahora a fondo. Se suponen que las reinfecciones son posibles y que probablemente son dictadas por dos variables: la exposición al virus y la duración de la inmunidad adquirida tras la primera infección.
Actualmente no es posible investigar las reinfecciones por SARS-CoV-2 ya que estamos en una fase temprana de la pandemia, sin embargo los coronavirus estacionales [causante del resfriado común] pueden servir como modelo de estudio [dada la similitud silogenética entre los mismos y el SARS-CoV-2]. Hay cuatro especies de coronavirus estacionales: HCoV-NL63, HCoV-229E, HCoV-OC43 y HCoV-HKU1. Todos están asociados con infecciones leves del tracto respiratorio, pero los cuatro virus son genética y biológicamente diferentes. Dos pertenecen al género Alphacoronavirus , y dos al género Betacoronavirus [mismo género del SARS-CoV-2]. Los virus usan diferentes moléculas receptoras para ingresar a una célula objetivo, y en función de la distribución del receptor, no todas ingresan al mismo tipo de células epiteliales en los pulmones. Dada esta variabilidad, los coronavirus estacionales son los grupos de virus más representativos del cual concluir características generales de los coronavirus en cuanto a denominadores comunes como la dinámica de la inmunidad y la susceptibilidad a la reinfección.
El objetivo de este estudio es investigar la duración de la protección del coronavirus contra las reinfecciones. Examinar las reinfecciones mediante la prueba del virus en el material respiratorio requiere la recolección de muestras respiratorias de voluntarios durante cada síntoma de resfriado común y, debido a que las reinfecciones pueden ser asintomáticas, también durante períodos sin síntomas, durante años seguidos. Este tipo de colecciones de muestras son difíciles de obtener. La alternativa es medir los aumentos en los anticuerpos contra un virus (serología), como un indicador de infección reciente. Esta opción es aplicable, ya que se ha determinado que los niveles de IgG para el coronavirus estacional: 1) solo aumentan después de una infección exitosa, 2) también aumentan con la infección asintomática, y 3) no aumentan después de un desafío viral fallido. Las muestras de sangre recolectadas a intervalos regulares en estudios de voluntarios durante décadas pueden servir posteriormente como material esencial para investigar la dinámica de la reinfección basada en una serología. En teoría, las pruebas de neutralización del virus pueden parecer el mejor ensayo serológico cuando se investiga la inmunidad protectora, sin embargo, existen limitaciones graves. Primero, no hay una línea celular que facilite la replicación de HCoV-HKU1 y, por lo tanto, no se pueden realizar pruebas de neutralización para este virus. En segundo lugar, las únicas cepas de virus cultivadas HCoV-229E y HCoV-OC43 disponibles son de la década de 1960 y están adaptadas al laboratorio, las cuales pueden no ser representantes adecuados de los virus en estado salvaje. En cambio, medir los niveles de anticuerpos dirigidos a proteínas virales usando, por ejemplo, ELISA, sí permitirá la prueba de reinfección para los 4 coronavirus estacionales. En este caso, se debe hacer una elección cuidadosa para el antígeno, considerando la compensación entre sensibilidad y reactividad cruzada en un ensayo serológico. Aunque la proteína Spike provoca anticuerpos neutralizantes, es la menos conservada dentro de una especie de coronavirus estacional, y disminuye la sensibilidad para detectar infecciones. En contraste, la proteína N, y específicamente su región C terminal (NCt), está significativamente más conservada y se ha encontrado como la proteína más inmunogénica, específica y sensible para controlar las infecciones por coronavirus estacionales [5–9].
Dinámica dela infección.
De un estudio de prospectivo realizado con hombres adultos (ver M&M, 10), se seleccionaron diez sujetos que participaron desde el inicio del estudio y al menos 10 años en seguimiento. Estos participantes no informaron ninguna enfermedad grave que pudiera haber influido en su inmunidad (ver descripción del estudio en M&M). El seguimiento de los sujetos con recolección y almacenamiento de sangre comenzó en 1985 y, dejando de lado una brecha en el seguimiento entre 1997 y 2003, continuó para la mayoría de los sujetos hasta el 2020 a intervalos regulares (cada 3 meses antes de 1989 y cada 6 meses después). El período acumulativo en el que los sujetos fueron seguidos continuamente (es decir, < 400 días de intervalo entre muestras consecutivas) totalizó más de 200 años-persona = 2473 meses. Al comienzo del estudio, la edad de los sujetos varió de 27 a 40 años; Al final del seguimiento, los sujetos tenían entre 49 y 66 años.
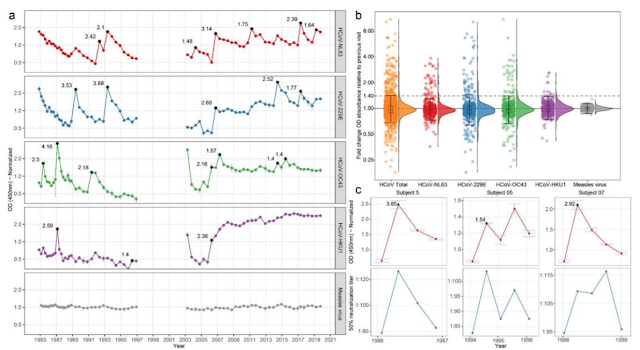
Figura 1 - Dinámica de anticuerpos para coronavirus estacionales y virus del sarampión en el tiempo. (a) Dinámica de anticuerpos del sujeto 9. Los puntos conectados indican intervalos de seguimiento < 400 días. Los asteriscos significan visitas clasificadas como infecciones, el número adyacente al asterisco describe el cambio de pliegue observado en la densidad óptica del ELISA. (b) Distribución de cambios de pliegue OD entre visitas secuenciales. Los gráficos muestran valores medios con el rango intercuartil (IQR), los picos se extienden hasta el rango después del cual las muestras se consideran valores atípicos (1.5 x IQR por debajo del primero o por encima del tercer cuantil). En rojo: HCoV-NL63; azul: HCoV-229E; verde: HCoVOC43; púrpura: HCoV-HKU1; naranja: coronavirus estacionales totales; gris: virus del sarampión. (c) La reactividad de anticuerpos a HCoV-NL63-NCt se asocia con la neutralización de HCoV-NL63. En rojo: dinámica de anticuerpos medida por ELISA; azul: disolución de neutralizadores.
Las infecciones por coronavirus se determinaron midiendo los cambios de pliegue en la densidad óptica (DO) en el reconocimiento de anticuerpos NCt entre dos visitas consecutivas (mostradas para un individuo [el 9] en la Fig. 1a y para todos sujetos en la figura complementaria (S1). Primero medimos la fluctuación natural entre visitas consecutivas en anticuerpos contra el virus del sarampión para los 10 sujetos suponiendo que el sarampión no ocurrió durante el seguimiento, ya que todos los sujetos fueron vacunados durante la infancia. Los cambios de pliegue en la OD del anticuerpo para el virus del sarampión variaron entre 0,85 y 1,28 (Fig. 1b). Luego se determinó un umbral para la infección por coronavirus utilizando la distribución del cambio de pliegue OD, suponiendo que durante la mayoría de los intervalos no se produjo infección por coronavirus y, por lo tanto, las infecciones aparecen como valores atípicos. La Figura 1b muestra que los aumentos de OD ≥1.4 veces fueron atípicos (Fig. 1b). Luego determinamos si estos criterios de infección serológica podrían deberse a enfermedades similares a la influenza (ILI) en el intervalo que precede directamente al aumento de anticuerpos. De hecho, la notificación de síntomas de ILI coincidió con un aumento ≥1.4 en los anticuerpos (prueba exacta de Fisher p = 0.031, tabla complementaria S1). Finalmente comparamos los resultados de ELISA de HCoV-NL63 con la disolución de neutralizadores para HCoV-NL63 en dos sujetos (# 5 y # 7, con tres infecciones). Las infecciones mostraron un aumento en la disolución de neutralizadores que acompañó al aumento ≥1,4 veces en los anticuerpos NCt (Fig. 1c).
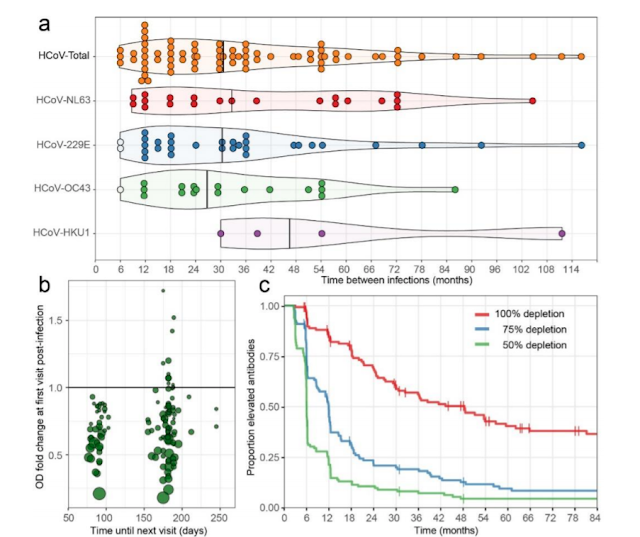
Un total de 132 eventos [infecciones], que van de 3 a 22 por sujeto, se clasificaron como infecciones por coronavirus (Tabla 1). Se encontraron tiempos medios de reinfección de 33 (IQR 18-60), 31 (IQR 15-42), 27 (IQR 21-49) y 46 (IQR 36-68) para HCoV-NL63, HCoV-229E, HCoV- OC43 y HCoV-HKU1, respectivamente, y 30 (IQR 18-54) meses para todos los virus combinados (Fig. 2a). No hubo diferencia estadísticamente significativa entre las longitudes de intervalo de infección de los virus individuales (prueba de Kruskal-Wallis, P = 0,74). En algunos casos, las reinfecciones ocurrieron tan pronto como los 6 meses (dos veces para HCoV-229E y una vez para HCoV-OC43) y los 9 meses (dos veces para HCoV-NL63). El tiempo de reinfección más frecuente observado fue de 12 meses. Para las reinfecciones que ocurrieron a los 6 meses no observamos reducción en los anticuerpos entre infecciones (Fig. 2a, círculos blancos), sin embargo, para los tiempos de reinfecciones > 6 meses se encontraron reducciones en los niveles de anticuerpos entre dos infecciones (visibles como picos en la Fig. 1a y Complementaria Fig. S1).
La capacidad de detectar reinfecciones a corto plazo está limitada por el intervalo de muestreo. Sin embargo, es importante destacar que no se observó reinfección en la primera visita de seguimiento posterior después de un intervalo de 3 meses (Fig. 2a). Observamos varias reinfecciones en visitas posteriores con un intervalo de 6 meses, lo que sugiere que no ocurren reinfecciones dentro de los 6 primeros meses. Para examinar más de cerca esto, también observamos cambios < 1.4 veces menor en los anticuerpos directamente después de una infección, siempre bajo el supuesto de que los niveles de anticuerpos que no disminuyen adecuadamente después de la infección pueden ser un signo de otra infección. Como se muestra en la figura 2b, solo se encontraron cambios por debajo de 1 después de la infección con cada muestreo de 3 meses y, por lo tanto, podemos concluir con seguridad que el punto de tiempo de confianza más temprano para la reinfección es de 6 meses.
Figura 2. Características de infección y reinfección, y dinámica de anticuerpos menguantes para coronavirus estacionales. (a) El intervalo de tiempo entre reinfecciones. Los puntos blancos indican reinfecciones para las cuales no se pudo observar una disminución intermedia en los niveles de anticuerpos. Las líneas verticales negras describen los tiempos medios de reinfección. (b) Cambios en los niveles de anticuerpos después de la infección en relación con la duración del intervalo de seguimiento. Cada círculo representa una infección. El eje x describe el tiempo hasta la próxima visita de seguimiento posterior a la infección. El eje y describe el cambio en el nivel de anticuerpos en la visita posterior. Los círculos más grandes representan una mayor relación de aumento en los niveles de anticuerpos en la infección inicial. La línea horizontal indica el límite entre aumentos (> 1.0) o disminuciones (< 1.0) en los niveles de anticuerpos en la próxima visita de estudio. (c) Curva de Kaplan-Meier que muestra disminución de anticuerpos después de la infección (100%, 75% y 50%), detalles en M&M.
Dinámica de anticuerpos después de la infección.
La inmunidad protectora puede durar menos que el tiempo medido hasta la reinfección porque una reinfección también requiere exposición a un virus. Por lo tanto, el decaimiento en los niveles de anticuerpos puede ser un mejor marcador de la inmunidad menguante. Aunque los anticuerpos contra la proteína N por sí mismos no son neutralizantes, pueden considerarse como un representante del total de anticuerpos (ver comparaciones con la neutralización Fig. 1c). Analizamos la dinámica de la disminución de los anticuerpos NCt después de la infección, calculando el tiempo hasta que se produjo un retorno del 50%, 75% o del total de los niveles de anticuerpos a la línea de base (niveles de anticuerpos previos a la infección). La mayoría de los pacientes perdieron el 50% de sus anticuerpos anti-proteína NCt después de 6 meses, el 75% después de un año, y regresaron completamente a los valores basales 4 años después de la infección (Fig. 2c y Fig. Suplementaria S1).
Infecciones simultáneas.
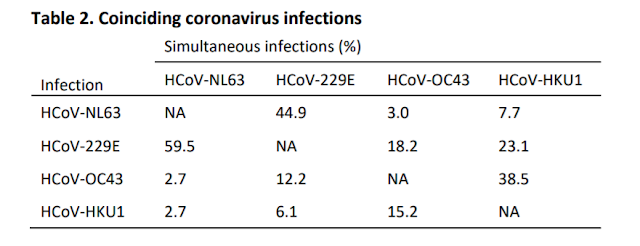
Aunque nuestras pruebas ELISA que usan la parte C-terminal de la proteína N se diseñaron con precaución para ser específicas para cada virus individual, no podemos descartar que ocurra un cierto grado de reactividad cruzada de anticuerpos. Por lo tanto, investigamos con qué frecuencia coincidían las infecciones, ya que la reactividad cruzada puede haber llevado a un etiquetado falso de las infecciones. Observamos que las infecciones simultáneas con un Alfacoronavirus (HCoV-NL63 o HCoV-229E) junto con Betacoronavirus (HCoV-HKU1 o HCoV-OC43) eran raras, sin embargo, vimos que las infecciones por los Betacoronavirus HCoV-OC43 y HCoV-HKU1 a menudo coincidían (38,5%, tabla 2). Asimismo, para los Alfacoronavirus, las infecciones por HCoV-229E coincidieron con las infecciones por HCoV-NL63 en el 59.5% de los casos, y viceversa en el 44.9% de los casos. Por lo tanto, existe el riesgo de que sobrestimásemos el número de infecciones y, por lo tanto, volvimos a analizar los datos con una definición más estricta de infección, que incluye solo el aumento de los anticuerpos más fuertes inducidos por un Betacoronavirus o Alphacoronavirus en un momento determinado. Según esta definición, todavía encontramos intervalos de infección comparables a los datos originales (Fig. S2 suplementaria), con intervalos mínimos de infección tan cortos como 6 meses y reinfecciones frecuentes a los 12 meses, aunque el número de reinfecciones obviamente se redujo.
Estudiando los anticuerpos comunes contra todos los coronavirus.
En teoría, los anticuerpos inducidos por infecciones por coronavirus pueden tener amplias características de reconocimiento en común. Para examinar esto, realizamos un ELISA adicional en los 10 sujetos, esta vez utilizando la proteína N completa de SARS-CoV-2, para permitir la detección de anticuerpos ampliamente reconocidos en coronavirus. Para excluir que la detección de este tipo de anticuerpos fueron falsos positivos debido a que fueron dirigidos a la etiqueta his en nuestras proteínas N de coronavirus, también realizamos un ELISA de control con una proteína de envoltura de VIH-1 etiquetada con his. Un sujeto (# 2) mostró un reconocimiento no específico, ya que también se reconoció la proteína VIH-1-SOSIP etiquetada con his (Figura complementaria S3). Otros dos sujetos mostraron anticuerpos ampliamente compartidos, muy probablemente inducidos por infecciones con Alphacoronavirus y Betacoronavirus durante el mismo intervalo (sujetos 9 y 10, Suplemento Fig. S3). Es de notar que no parece que estos anticuerpos compartidos sean buenos protectores ya que las infecciones por HCoV-NL63, HCoV-229E y HCoVOC43 ocurrieron en presencia de estos anticuerpos (Figura complementaria S1).
Infecciones por coronavirus según las estaciones del año.
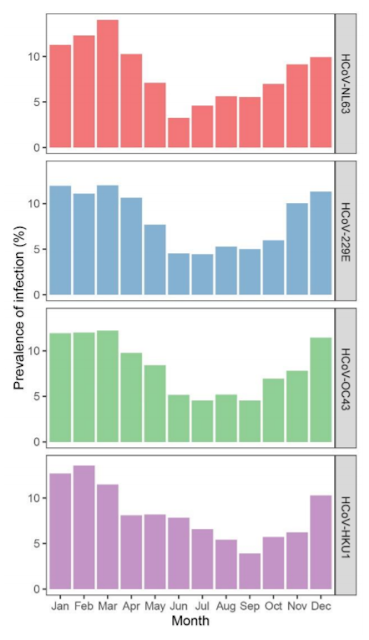
Hasta la fecha, no está claro si el SARS-CoV-2 compartirá el mismo pico de prevalencia invernal que se observa en los coronavirus estacionales en países no ecuatoriales. Sin embargo, es importante considerar que la preferencia invernal de los coronavirus estacionales solo se ha determinado analizando muestras respiratorias de personas que experimentaron la enfermedad. Por lo tanto, el muestreo y el almacenamiento dependen de los síntomas y no del protocolo de estudio. Si la propagación del coronavirus continúa sin disminuir en verano, sin embargo, las personas rara vez muestran síntomas y, por lo tanto, no se toman muestras, las infecciones permanecerán sin ser detectadas. Nuestro estudio serológico es único porque evita este sesgo de muestreo. Los Países Bajos tienen un clima templado típico, y nuestras muestras de estudio fueron recolectadas a intervalos regulares. Las muestras de cada sujeto se distribuyeron aleatoriamente durante todo el año y, debido al régimen de visitas de 3 o 6 meses, se recolectaron muestras durante todas las estaciones. En consecuencia, podemos por primera vez visualizar la estacionalidad de las infecciones por coronavirus de manera imparcial. Estimamos la prevalencia de inicio de infección para cada mes para todas las infecciones detectadas en este estudio (ver los métodos suplementarios para detalles de análisis). Como se muestra en la Fig. 4, los meses de primavera y verano de mayo, junio, julio, agosto y septiembre muestran la prevalencia más baja de infecciones para los cuatro coronavirus estacionales (prueba de Wilcoxon de rango con signo, p = 0.005).
Fig. 4 Estacionalidad de las infecciones. Prevalencia de infección de los cuatro coronavirus estacionales en diferentes meses. La prevalencia por mes se muestra como un porcentaje del número total de infecciones por coronavirus.
DISCUSIÓN
Mostramos, por primera vez, que las reinfecciones por todos los coronavirus estacionales ocurren de manera natural. La mayoría de las reinfecciones ocurrieron en 3 años. Sin embargo, este intervalo de tiempo entre infecciones no indica que la inmunidad protectora de un individuo dure el mismo período de tiempo, ya que la reinfección también depende de la reexposición. De hecho, según los intervalos mínimos de infección y la dinámica de disminución de anticuerpos que observamos, la inmunidad protectora puede durar tan poco como 6 a 12 meses. Recientemente Kissler et al. modeló la inmunidad protectora y la dinámica de reinfección de los HCoV-OC43 y HCoV-HKU1 y estimó un período de inmunidad protectora de 45 semanas. Nuestro estudio serológico confirma esta predicción. Cuando vemos nuestros hallazgos a la luz de las acciones de control actuales tomadas para el SARS-CoV-2, queda claro que el riesgo de reinfección del coronavirus es clave para la política de salud pública. Aquí revelamos un riesgo de que en el futuro cercano, las pruebas basadas en serología que miden infecciones previas para el SARS-CoV-2 que usan la proteína N puedan tener un uso limitado si esa infección se produjo >1 año antes del muestreo. Nuestro estudio también muestra que la inmunidad colectiva puede ser un desafío debido a la rápida pérdida de la inmunidad protectora. Recientemente se sugirió que las personas recuperadas deberían recibir el llamado "pasaporte de inmunidad" que les permitiría relajar las medidas de distanciamiento social y proporcionar a los gobiernos datos sobre los niveles de inmunidad de rebaño en la población. Sin embargo, como la inmunidad protectora puede perderse a los 6 meses después de la infección, la posibilidad de alcanzar la inmunidad funcional del rebaño por infección natural parece muy poco probable.
Notamos que tres sujetos portaban anticuerpos que reconocían la proteína N del SARS-CoV-2 en ciertos puntos de tiempo. Es poco probable que hayan sido infectados con un virus similar al SARS-CoV-2 en 1985 (sujeto # 10), 1992 (sujeto # 2) o 2006 (sujeto # 9), y por lo tanto, sugerimos que estos anticuerpos ampliamente compartidos para hacer frente a los coronavirus han sido inducidos por infecciones coincidentes de un alfa y un betacoronavirus (en nuestros sujetos HCoV-HKU1 y HCoV-NL63). Para explorar este hallazgo, observamos la distancia genética y, en consecuencia, las diferencias de aminoácidos en la proteína estructural de los diversos coronavirus (tabla complementaria S2). La proteína N del SARS-CoV-2 tiene solo un 32% y 34% de coincidencia a nivel de aminoácidos con la proteína N del HCoV-OC43 y el HCoV-HKU1 respectivamente, y solo un 26% y 24% de identidad con HCoV-NL63 y HCoV-229E. De manera similar, la distancia entre el Alphacoronavirus y la proteína N del Betacoronavirus es grande (solo 24% a 26% de identidad entre aminoácidos). Aún así, no podemos excluir la presencia de epítopos conservados (conformacionales) en la proteína N del HCoV-HKU1 y el HCoV-NL63 que pueden dar como resultado una respuesta de anticuerpos de acción más amplia, debido a la exposición simultánea en infecciones concurrentes. Se requiere una evaluación adicional, que incluya más sujetos, para la confirmación.
No pudimos secuenciar el genoma del virus durante la infección. En teoría, la variación de la cepa podría jugar un papel en la susceptibilidad a la reinfección. HCoV-NL63, HCoV-OC43 y HCoV-HKU1 muestran diferentes grupos genéticos cocirculantes [4,14,15]. La situación es aún más complicada para HCoV-229E. Sylvia Reed ha demostrado que la reinfección experimental de voluntarios no tiene éxito cuando se usa la misma cepa de HCoV-229E, pero tiene éxito cuando se usan cepas heterólogas. Curiosamente, no se conocen subtipos genéticos importantes para HCoV-229E [17,18]. Como HCoV-NL63 no se conocía en la década de 1980, y las características del cultivo diferían entre las cepas HCoV-229E de Reed, las llamadas cepas heterólogas en realidad pueden haber sido HCoVNL63 [16,19]. Un estudio sobre inmunidad protectora idealmente permitiría la secuenciación de las cepas de reinfección de material respiratorio; sin embargo, esto es intratable en un estudio de infección natural porque la eliminación del virus en las reinfecciones puede ser tan breve como un día, y los esquemas de muestreo respiratorio serían extremadamente engorrosos para los voluntarios. Otra limitación del estudio es que los sujetos en nuestro estudio eran todos hombres. Para COVID-19, y también HCoV-NL63, los hombres tienen una mayor incidencia de la enfermedad [20] y, por lo tanto, es interesante determinar la dinámica de la inmunidad protectora también en una cohorte de mujeres sanas. En conclusión, todos los coronavirus humanos estacionales tienen poco en común, aparte de causar un resfriado común. Aún así, todos parecen inducir una inmunidad de corta duración con una rápida pérdida de anticuerpos. Este bien puede ser un denominador general para los coronavirus humanos.
Referencias
1. Callow, K. A. Efecto de la inmunidad humoral específica y algunos factores no específicos sobre la resistencia de voluntarios para la infección por coronavirus respiratorio. Epidemiology & Infection 95, 173-189 (1985).
2. van der Hoek, L. y col. Carga de la enfermedad debida a las infecciones por coronavirus NL63 humano y periodicidad de la infección. Journal of Clinical Virology 48, 104-108 (2010).
3. van der Hoek, L. Coronavirus humanos: ¿qué causan? Terapia antiviral 12, 651 (2007).
4. Pyrc, K. y col. Estructura mosaica del coronavirus humano NL63, mil años de evolución.
Journal of molecular biology 364, 964–973 (2006).
5. Dijkman, R. y col. El dominio de las infecciones por coronavirus humano OC43 y NL63 en lactantes. Journal of Clinical Virology 53, 135–139 (2012).
6. Cesantía, E. G. et al. Desarrollo de un inmunoensayo de coronavirus humano basado en nucleocápsides y estimaciones de individuos expuestos al coronavirus en una población metropolitana de EE. UU. Clínico y Vaccine Immunology 15, 1805-1810 (2008).
7. Sastre, P. y col. Diferenciación entre coronavirus humanos NL63 y 229E usando una novela
ensayo inmunosorbente ligado a enzimas sandwich de doble anticuerpo basado en un monoclonal específico anticuerpos Clinical and Vaccine Immunology 18, 113–118 (2011).
8. Dijkman, R. y col. Coronavirus humano NL63 y seroconversión 229E en niños. Journal of Clinical
Microbiology 46, 2368–2373 (2008).
9. Lehmann, C. y col. Una línea de inmunoensayo que utiliza proteínas nucleocápsides recombinantes para la detección de anticuerpos contra coronavirus humanos. Microbiología diagnóstica y enfermedades infecciosas 61, 40–48 (2008).
10. van Bilsen, W. P. H. y col. Tendencias divergentes en la incidencia del VIH versus otras enfermedades de transmisión sexual. Infecciones en HSH VIH negativos en Amsterdam. SIDA 34, (2020).
11. Gaunt, E. R., Hardie, A., Claas, E. C. J., Simmonds, P. y Templeton, K. E. Epidemiología y clínica presentaciones de los cuatro coronavirus humanos 229E, HKU1, NL63 y OC43 detectados en
años utilizando un nuevo método de PCR multiplex en tiempo real. Revista de microbiología clínica 48, 2940–2947 (2010).
12. Kissler, S. M., Tedijanto, C., Goldstein, E., Grad, Y. H. y Lipsitch, M. Proyectando la transmisión
dinámica del SARS-CoV-2 durante el período pospandémico. Ciencia (2020) doi: 10.1126 / science.abb5793.
13. Bendavid, E. y col. COVID-19 Anticuerpo Seroprevalencia en el Condado de Santa Clara, California. medRxiv 2020.04.14.20062463 (2020) doi: 10.1101 / 2020.04.14.20062463.
14. Woo, P. C. Y. y col. El análisis comparativo de 22 genomas de coronavirus HKU1 revela una novela genotipo y evidencia de recombinación natural en coronavirus HKU1. Revista de virología 80,
7136–7145 (2006).
15. Lau, S. K. P. y col. La epidemiología molecular del coronavirus humano OC43 revela la evolución de diferentes genotipos a lo largo del tiempo y la reciente aparición de un nuevo genotipo debido a la naturaleza recombinación Journal of virology 85, 11325–11337 (2011).
16. Reed, S. E. El comportamiento de aislamientos recientes de coronavirus respiratorio humano in vitro y en voluntarios: evidencia de heterogeneidad entre las cepas relacionadas con 229E. Revista de virología médica 13, 179-192 (1984).
17. Chibo, D. y Birch, C. Análisis de los genes de la espiga y nucleoproteína del coronavirus humano 229E demuestra la deriva genética entre cepas cronológicamente distintas. Revista de virología general 87, 1203-1208 (2006).
18. Farsani, S. M. J. y col. Las primeras secuencias completas del genoma de aislados clínicos de humanos coronavirus 229E. Virus Genes 45, 433–439 (2012).
19. van der Hoek, L., Pyrc, K. y Berkhout, B. Coronavirus humano NL63, un nuevo virus respiratorio. FEMS microbiology reviews 30, 760–773 (2006).
20. Kiyuka, P. K. y col. Coronavirus humano NL63 epidemiología molecular y patrones evolutivos en
Kenia costera rural. El diario de enfermedades infecciosas 217, 1728-1739 (2018).